科学家25年追逐线虫大脑模拟,从失败到曙光初现。这不仅解锁微小生命之谜,更为人类智能进化点燃希望。
25年来,科学家们一直在尝试用计算机重现我们所知的最小大脑,却总是铩羽而归。如今,技术终于成熟,让这个梦想触手可及。如果能精确模拟人类大脑,那将是何等震撼?它能让我们突破生物桎梏,释放潜能,开辟智能、创造力和探索的新天地。这或许是人类进化的下一站,让我们的思维与记忆摆脱肉体的束缚。
然而,这条路还很长。人类大脑拥有千亿神经元,彼此通过高达千万亿的突触相连。要逆向破解这个庞大网络,需要的计算能力远超当下。为了找到突破口,科学家们把目光转向了更简单的生物。而最简单的选择,莫过于仅有300个神经元的线虫Caenorhabditis elegans。
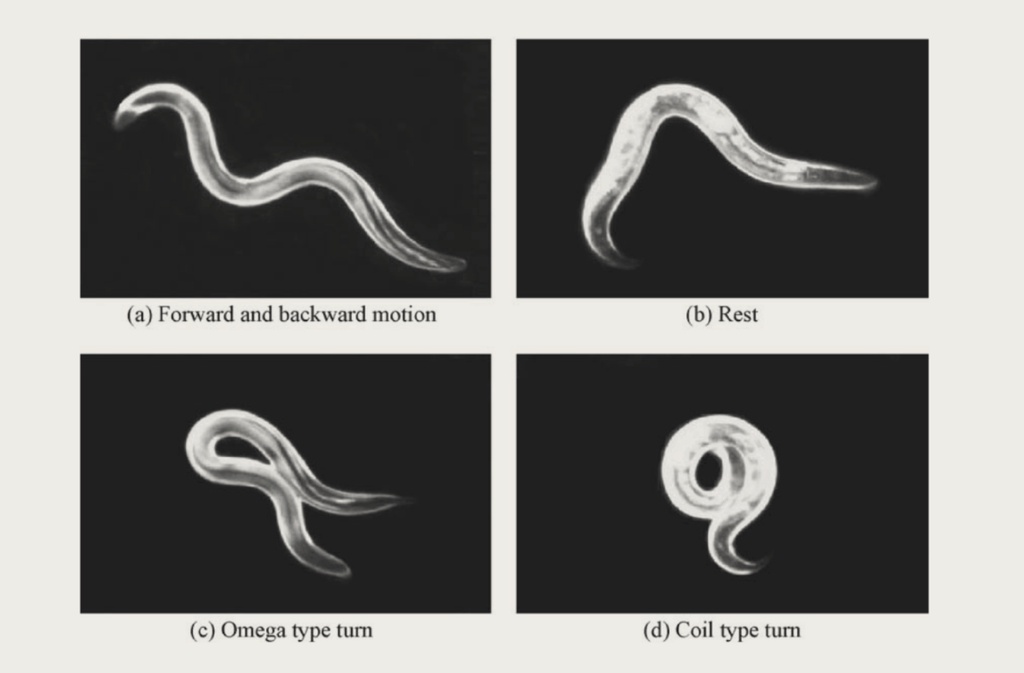
C. elegans的运动模式
过去25年,研究者们尝试过各种方法模拟C. elegans的大脑,却收效甚微。但现在,我坚信这项任务不仅可能,更是必须完成的。技术进步让我们站在了新的起点上。
早在1970年代,生物学家Sydney Brenner就看中了C. elegans的潜力。它的简单结构和小巧体型,使它成为实验室的宠儿。1986年,Brenner团队的科学家John C. White绘制了C. elegans大脑的神经连接图,也就是如今所谓的“连接组”。随着计算机的普及,其他人开始接力。1991年,Ernst Neibur和Paul Erdös率先推出了线虫运动的生物物理模型。到了90年代末,来自俄勒冈州大学和日本的两支团队分别提出了更宏大的计划,试图利用White的成果打造新模型。可惜,这两个项目都未能起飞。
2004年,广岛大学的Virtual C. elegans项目迈出了一步。他们发表了两篇论文,展示了一个能模拟线虫运动控制的模型。虚拟线虫能对头部刺激做出反应,但也仅此而已。这甚至算不上真正的模拟,因为研究者缺乏神经元间连接的精确电学特性数据。他们转而用机器学习填补空白,让模拟线虫的表现接近真实,但这与生物现实仍有距离。这种困境,在后来的尝试中反复出现。
到了2010年代初,进展依然停滞。模拟线虫运动还有人在做,但大脑模拟却毫无起色。直到2010年元旦,工程师Giovanni Idili在推特上喊话Whole Brain Catalogue,立下新年目标:“模拟完整的C. elegans大脑(302个神经元)!”U.C. San Diego的神经科学研究生Stephen Larson看到了这条推文。到了8月,他在学术会议上抛出了这个想法。2011年初,他和Idili组建团队,启动了OpenWorm项目——一群分散的学者合力打造一个真实、开源的C. elegans模型。
那是个令人兴奋的年代,微小大脑的模拟成了热门。几年间,OpenWorm接连发表论文,更新模型。2013年,他们在巴黎开了首场会议,还登上了《大西洋月刊》,标题乐观地写道:“这只虚拟蠕虫是奇点的前兆吗?”与此同时,MIT的David Dalrymple也在推进一个名为Nemaload的项目。OpenWorm多用死线虫数据,而Dalrymple想借助新兴的光遗传学研究活体,用光控制神经元,采集大脑对各种状态的反应数据。他曾在2011年的LessWrong评论中大胆预测:“如果2020年这还是难题,我会非常惊讶。”
如今是2025年,线虫大脑模拟仍是未解之谜。Dalrymple在2012年放弃了Nemaload。OpenWorm虽未解散,但过去十年因数据不足,进展寥寥。偶尔有新模拟问世,但仍依赖大量假设。我们比2010年代强些,数据更丰富,工具更先进,可距离完整模拟还差得远。到底哪里出了问题?为何25年未能模拟这最简单的大脑?更重要的是,我为何相信现在能成功?
在找原因前,我们得先问:模拟大脑到底意味着什么?学术界对“模拟”这词颇有争议。比如2013年,Henry Markram拿到欧盟10亿欧元,宣称要模拟人类大脑,可这计划当时就被认为不切实际。最终项目受挫,目标未达,给脑模拟研究蒙上阴影。从那以后,设定清晰、可行的目标变得尤为重要。
我眼中的好模拟,不仅要复制大脑功能,还要能预测真实大脑的行为。就像把虚拟线虫放在虚拟培养基上,它的反应得和真线虫一模一样。戳它、照光,它得有真实反应,且长时间保持一致,不出错。去年,127家机构合作完成了果蝇Drosophila melanogaster的完整连接组,包含14万多神经元,是继C. elegans后的第二大成果。项目FlyWire点燃了模拟热潮。研究者Philip Shiu测试糖感知神经元时,模型预测果蝇会伸出喙管,和真果蝇一致。这模型还成功预测了味觉、梳理和运动的神经模式。
但这不算真正模拟。Shiu自己也说,模型过于简化,假设太多。就像OpenWorm一样,FlyWire缺的是神经结构与功能的精准数据。连接组像张地图,告诉你神经元怎么连,却不说连得怎么样。要模拟大脑,我们得知道每个神经元的电学特性,比如突触强弱、膜电容,甚至学习时阈值的变化。只靠静态连接组,模拟学不会东西,也就无法像真生物那样行动。
获取活体大脑的动态特性,比弄清结构难得多。电子显微镜能拍出高分辨率图像,可只能用在切片组织上,无法捕捉活脑反应。几十年来,唯一办法是“膜片钳”技术,精确但费力,一次最多测三个神经元。2013年时,能解锁神经元内部奥秘的工具要么不存在,要么不成熟,C. elegans模拟就卡在这儿。
正当线虫研究冷却时,细胞观测技术却在突飞猛进。光学显微镜的进步,让我们能快速拍下活细胞的清晰影像。传统体视显微镜用小孔挡住杂光,成像虽清楚却慢。新兴的光片显微镜用激光片照亮二维截面,速度快、对组织损伤小。2010年代初,超分辨率显微镜打破了光的衍射极限,分辨率达100纳米以下。2014年发明的DiSPIM用双镜头组,成像速度翻倍。2015年,哥伦比亚大学推出SCAPE,用倾斜光片单镜头扫描更快,特别适合追踪神经活动。
还有一大突破是荧光报告蛋白。当神经元放电,钙离子涌入,钙成像成了观测利器。2013至2015年,珍妮莉亚研究园的GCaMP6系列问世,比前辈更亮更敏感,成了活体神经成像标配。如今,DiSPIM和SCAPE结合钙成像,能实时看清C. elegans大脑全貌。接下来,得大量采集数据。White在1986年拼凑了五只线虫的连接组,但个体间神经元大小、电荷容量都不同。要彻底搞懂,得测几千只线虫。
有了数据怎么办?机器学习帮了大忙。C. elegans大脑虽复杂,可300个神经元对现代算法不算啥。用符号回归技术,我们能从神经活动数据推导出每个神经元的电容和突触强度,类似膜片钳的结果,但直接来自全脑数据。
我不是说模拟C. elegans很简单。突触外信号、神经元形态变化,这些都可能成难题。但现代技术日新月异,我相信这不再是幻想。如果想模拟人类这样的大脑,这一步更是绕不过。光片显微镜虽强,可光只能穿透不到1毫米的组织。现在,能全脑成像的只有C. elegans、斑马鱼幼体和果蝇。通过研究这些小生物,我们能找到预测神经活动的新法子,逐步攻克更大的大脑。
我的研究是用显微镜、荧光蛋白和机器学习,打造一个科学的C. elegans模拟,建一套可复制的蓝图。这不仅是科学壮举,更能揭示大脑结构与动态的联系。路还长,但现在是最好的起点,得快步走,目标明确,一步步来。
